Proteínas são macromoléculas biológicas constituídas por uma ou mais cadeias de aminoácidos. As proteínas estão presentes em todos os seres vivos e participam em praticamente todos os processos celulares, desempenhando um vasto conjunto de funções no organismo, como a replicação de ADN, a resposta a estímulos e o transporte de moléculas. Muitas proteínas são enzimas que catalisam reações bioquímicas vitais para o metabolismo. As proteínas têm também funções estruturais ou mecânicas, como é o caso da actina e da miosina nos músculos e das proteínas no citoesqueleto, as quais formam um sistema de andaimes que mantém a forma celular. Outras proteínas são importantes na sinalização celular, resposta imunitária e no ciclo celular. As proteínas diferem entre si fundamentalmente na sua sequência de aminoácidos, que é determinada pela sua sequência genética e que geralmente provoca o seu enovelamento numa estrutura tridimensional específica que determina a sua atividade.
Ao contrário das plantas, os animais não conseguem sintetizar todos os aminoácidos de que necessitam para viver. Os aminoácidos que o organismo não é capaz de sintetizar por si próprio são denominados aminoácidos essenciais e devem ser obtidos pelo consumo de alimentos que contenham proteínas, as quais são transformadas em aminoácidos durante a digestão. As proteínas podem ser encontradas numa ampla variedade de alimentos de origem animal e vegetal. A carne, os ovos, o leite e o peixe são fontes de proteínas completas. Entre as principais fontes vegetais ricas em proteína estão os legumes, principalmente o feijão, as lentilhas, a soja ou o grão-de-bico. A grande maioria dos aminoácidos está disponível na dieta humana, pelo que uma pessoa saudável com uma dieta equilibrada raramente necessita de suplementos de proteínas. A necessidade é também maior em atletas ou durante a infância, gravidez ou amamentação, ou quando o corpo se encontra em recuperação de um trauma ou de uma operação. Quando o corpo não recebe as quantidades de proteínas necessárias verifica-se insuficiência e desnutrição proteica, a qual pode provocar uma série de doenças, entre as quais atraso no desenvolvimento em crianças ou kwashiorkor.
Uma proteína contém pelo menos uma cadeia polímérica linear derivada da condensação de aminoácidos, ou polipeptídeo. Os resíduos individuais de aminoácidos estão unidos entre si através de ligações peptídicas. A sequência dos resíduos de aminoácidos em cada proteína é definida pela sequência de um gene, a qual está codificada nocódigo genético. Durante ou após o processo de síntese, os resíduos de uma proteína são muitas vezes alterados quimicamente através de modificação pós-traducional, a qual modifica as propriedades físicas e químicas das proteínas, o seu enovelamento, estabilidade, atividade e, por fim, a sua função. Nalguns casos as proteínas têm anexados grupos não peptídicos, os quais são denominados cofatores ou grupos prostéticos. As proteínas podem também trabalhar em conjunto para desempenhar determinada função, agrupando-se em complexos proteicos. As proteínas podem ser purificadas a partir de outros componentes celulares recorrendo a diversas técnicas, como a precipitação, ultracentrifugação, eletroforese e cromatografia. Entre os métodos usados para estudar a estrutura e funções das proteínas estão a imuno-histoquímica, mutagénese sítio-dirigida, ressonância magnética nuclear e espectrometria de massa.
Índice
[esconder]Nutrição[editar | editar código-fonte]
As proteínas são nutrientes essenciais ao corpo humano.[1] Enquanto a maior parte dos microorganismos e das plantas são capazes de biosintetizar todos os vinte aminoácidos-padrão, os animais, incluindo os seres humanos, necessitam de obter alguns desses aminoácidos a partir da dieta alimentar.[2] Isto deve-se à ausência nos animais de algumas enzimas-chave que têm como função sintetizar esses aminoácidos. Os aminoácidos que o organismo não é capaz de sintetizar por si próprio são denominados aminoácidos essenciais. Os animais podem obter aminoácidos através do consumo de alimentos que contenham proteínas. As proteínas ingeridas são transformadas em aminoácidos através da digestão, a qual envolve a desnaturação da proteína através da exposição ao ácido e à hidrólise por parte de enzimas denominadasproteases. Alguns dos aminoácidos ingeridos são usados para a biosíntese proteica, enquanto outros são convertidos em glicose, através de gliconeogénese, ou entram no ciclo do ácido cítrico.[3]
Além de constituírem a fundação dos tecidos do corpo, as proteínas são também uma fonte de energia. Enquanto fonte de energia, contêm 4 kcal por grama, valor semelhante aos hidratos de carbono, mas diferente dos lípidos, os quais contêm 9 kcal por grama. Durante a digestão, as proteínas são separadas no estômago em cadeiaspolipeptídicas mais pequenas através da ação do ácido clorídrico e da protease. Isto é essencial para a síntese dos aminoácidos essenciais que não podem ser biossintetizados pelo corpo.[5]
Os aminoácidos essenciais são a leucina, isoleucina, valina, lisina, treonina, triptófano, metionina, fenilalanina e histidina. Os aminoácidos não essenciais são a alanina,asparagina, ácido aspártico e ácido glutâmico. Os aminoácidos condicionalmente essenciais são a arginina, cisteína, glutamina, glicina, prolina, serina e tirosina.[6] Os aminoácidos encontram-se em diversas fontes alimentares de origem animal, como a carne, leite, peixe e ovos. As proteínas estão também disponíveis através de diversas fontes vegetais: cereais integrais, legumes, incluindo os secos, soja, fruta nozes e sementes. Os vegetarianos e vegans podem obter os aminoácidos essenciais necessários através da ingestão de diversas proteínas vegetais.[6]
Função das proteínas no corpo[editar | editar código-fonte]
As proteínas são nutrientes essenciais ao crescimento e manutenção do corpo humano.[7] Com a exceção da água, as proteínas são as moléculas mais abundantes no corpo, sendo o principal componente estrutural de todas as células, particularmente dos músculos.[8][9] As proteínas são também usadas em membranas, como é o caso dasglicoproteínas. Depois de serem repartidas em aminoácidos, são usadas como precursores do ácido nucleico, coenzimas, hormonas, resposta imunitária, reparação das células e outras moléculas essenciais para a vida.[10] As proteínas são ainda fundamentais para a formação de células sanguíneas.[11] Acredita-se que as proteínas aumentem o desempenho atlético.[12] Os aminoácidos são usados na produção de tecido muscular e na reparação de tecido danificado. As proteínas só são usadas como fonte de energia quando os recursos de hidratos de carbono e lipídos no corpo diminuem.[13]
Fontes alimentares[editar | editar código-fonte]
As proteínas podem ser encontradas numa ampla variedade de alimentos. A combinação mais adequada de fontes de proteína para cada pessoa depende da região, da acessibilidade, do custo económico, do tipo de aminoácidos e do equilíbrio nutricional, assim como do próprio paladar. Embora alguns alimentos sejam fontes ricas em determinados aminoácidos, o seu valor para na nutrição humana é limitado devido à sua pouca digestibilidade, a fatores antinutricionais, à elevada quantidade de calorias, ao colesterol ou à densidade mineral.[14]
A carne, os ovos, o leite e o peixe são fontes de proteínas completas.[15] Entre as fontes vegetais ricas em proteína estão os legumes,nozes, sementes e fruta. Os legumes têm maior concentração de aminoácidos e são fontes mais completas de proteína do que os cereais e os cereais integrais. Entre os alimentos vegetarianos com concentração de proteínas superior a 7% estão a soja, lentilhas,feijão vermelho e branco, feijão-frade, feijão-da-china, grão-de-bico, feijão-verde, tremoço, amêndoa, castanha-do-pará, cajueiro, noz-pecã e sementes de sésamo, deabóbora, de algodão e de girassol.[16]
Entre os alimentos de base que constituem uma fonte pobre em proteínas estão raízes e tubérculos como o inhame, mandioca e batata-doce, os quais contêm apenas entre 0 e 2% de proteínas. A fruta, embora seja rica noutros nutrientes essenciais, é uma fonte relativamente pobre de aminoácidos. A banana-da-terra é também pobre em proteínas. Para uma alimentação saudável, os alimentos básicos com baixo teor de proteína devem ser complementados com alimentos com proteínas completas e de qualidade, sobretudo durante o desenvolvimento das crianças.[17][18][19]
| Fonte alimentar | Lisina | Treonina | Triptófano | Aminoácidos com enxofre |
|---|---|---|---|---|
| Legumes | 64 | 38 | 12 | 25 |
| Cereais (incl. integrais) | 31 | 32 | 12 | 37 |
| Nozes e sementes | 45 | 36 | 17 | 46 |
| Fruta | 45 | 29 | 11 | 27 |
| Animal | 85 | 44 | 12 | 38 |
A grande maioria dos aminoácidos está disponível na dieta humana, pelo que uma pessoa saudável com uma dieta equilibrada raramente necessita de suplementos de proteínas.[6][20] Os aminoácidos mais limitados são a lisina, a treonina e os aminoácidos com enxofre.[21] A tabela em anexo mostra os mais importantes grupos alimentares que constituem fontes de proteínas, sob uma perspetiva mundial. Também lista o desempenho de cada grupo enquanto fonte dos aminoácidos mais limitados, em valores de miligramas de aminoácido limitado por cada grama de proteína total nesse alimento.[22]
Necessidades dietéticas[editar | editar código-fonte]
Existe um debate considerável sobre as necessidades relativas ao consumo de proteínas.[23][24] A quantidade de proteínas necessária na dieta de determinada pessoa é determinada em grande parte pelo consumo total de energia e hidratos de carbono, pela necessidade do corpo de nitrogénio e aminoácidos essenciais, composição e massa corporal, taxa de crescimento, nível de atividade física e presença de lesões ou doenças.[25][26][27] A atividade física elevada e o aumento da massa muscular aumentam a necessidade de proteínas. A necessidade é também maior durante a infância, gravidez ou amamentação, ou quando o corpo se encontra em recuperação de um trauma ou de uma operação.[28]
| Grupo | Valor diário recomendado (AESA)[29] |
|---|---|
| Adultos e idosos | 0,83 g/kg/dia |
| Recém-nascidos, crianças e adolescentes | 0,83 — 1,31 g/kg/dia |
| Grávidas | acréscimo de: 1º trimestre: 1 g/dia 2º trimestre: 9 g/dia 3º trimestre: 21 g/dia |
| Lactantes | acréscimo de: primeiros seis meses: 19 g/dia meses seguintes: 13 g/dia |
De acordo com os valores de referência de ingestão de proteínas da Autoridade Europeia para a Segurança Alimentar, os adultos, incluindo idosos, devem ingerir 0,83 g de proteína por dia por cada quilograma de peso corporal; os recém-nascidos, crianças e adolescentes devem ingerir entre 0,83 e 1,31 g/kg/dia, dependendo da idade. As grávidas devem ingerir valores suplementares de proteína: 1 g, 9g e 28g suplementares por dia durante o primeiro, segundo e terceiro trimestres, respetivamente. As lactantes devem também ingerir valores suplementares: 19 g por dia durante os primeiros seis meses de amamentação e 13 g por dia a partir dos seis meses.[29] De acordo com as recomendações norte-americanas e canadianas, as mulheres com idade entre 19 e 70 anos necessitam de consumir 46 g de proteínas por dia, enquanto os homens no mesmo intervalo etário necessitam de consumir pelo menos 56 g de proteínas por dia.[30] O valor geralmente recomendado para o consumo diário é de 0,8 g de proteínas por cada quilograma de massa corporal.[23] No entanto, esta recomendação baseia-se nas necessidades estruturais, sem considerar o uso de proteínas no metabolismo energético, pelo que se adequa a uma pessoa relativamente sedentária. [23][25] Diversos estudos têm concluído que as pessoas ativas e os atletas possam exigir um consumo superior de proteínas, devido ao aumento da massa muscular e da sudação, e da maior necessidade de proteínas enquanto fonte de energia e reparação do corpo.[23][24][25] Para estes casos, os valores sugeridos têm oscilado entre 1,6 g/kg e 1.8 g/kg.[24]
Consumo excessivo e insuficiência[editar | editar código-fonte]
Para compensar as variações na ingestão de proteínas ao longo do dia, ou em casos de emergência em que a ingestão de proteínas é temporariamente alta ou baixa, o corpo tenta equilibrar os níveis de proteínas recorrendo a uma reserva de curta duração.[31] No entanto, o corpo é incapaz de armazenar o excesso de proteínas a longo prazo. As proteínas são digeridas em aminoácidos que entram na corrente sanguínea. Os aminoácidos em excesso são convertidos pelo fígado em moléculas úteis, num processo denominado desaminação. A desaminação converte onitrogénio dos aminoácidos em amónia, a qual é por sua vez convertida pelo fígado em ureia durante o ciclo da ureia. A ureia é depois excretada pelos rins.[25][32]
O consumo excessivo de proteínas provoca também o aumento da excreção de cálcio na urina, o que se pensa ser devido ao desequilíbrio no pH, agravando o risco da formação de cálculos no sistema urinário.[31]Um estudo epidemiológico de 2006 não verificou a existência de qualquer relação entre o consumo total de proteína e a pressão arterial, embora tenha verificado uma relação inversa entre o consumo de proteína vegetal e a pressão arterial.[33]
Quando o corpo não recebe as quantidades de proteínas necessárias verifica-se insuficiência e desnutrição proteica, a qual pode provocar uma série de doenças, entre as quais atraso no desenvolvimento em crianças, kwashiorkor, pigmentação avermelhada do cabelo e da pele, fígado gorduroso, diarreia, dermatose e diminuição na contagem de linfócitos T, o que aumenta o risco de infeções secundárias.[34][35] A desnutrição proteica é relativamente comum à escala mundial, tanto em adultos como crianças, e é responsável por cerca de seis milhões de mortes anualmente. Nos países desenvolvidos, esta doença verifica-se predominantemente em idosos ou em hospitais, geralmente associada a outras doenças.[36]
Dietas proteicas[editar | editar código-fonte]
As dietas de alto teor proteico podem ajudar a perder peso ao fazer com que a pessoa se sinta cheia mais rapidamente. Na maioria das pessoas saudáveis, este tipo de dietas não apresenta riscos para a saúde, sobretudo se for seguida durante um curto período de tempo. No entanto, os riscos a longo prazo estão ainda em estudo. O recurso prolongado a este tipo de dieta, geralmente associadas com a limitação do consumo de hidratos de carbono, pode causar insuficiências nutricionais e falta de fibras, o que por sua vez provoca dores de cabeça e obstipação. Algumas destas dietas baseiam-se no aumento do consumo de carne vermelha e lacticínios gordos, o que aumenta o risco de doenças cardiovasculares. As escolhas mais saudáveis para uma dieta de alto teor proteico incluem proteína de soja, feijão, nozes, peixe, aves de criação sem pele, carne de porco e lacticínios magros, devendo ser evitadas as carnes vermelhas e processadas.[37]
Bioquímica[editar | editar código-fonte]
A maior parte das proteínas consiste em polímeros lineares formados a partir de um máximo de 20 L-α-aminoácidos. Todos os aminoácidos proteinogénicos têm em comum diversas características estruturais, entre as quais um carbono alfa, ao qual estão quimicamente ligados um grupo de aminas, um grupo de ácido carboxílico e uma cadeia lateral variável. Apenas a prolina difere desta estrutura básica.[38] As cadeias laterais dos aminoácidos comuns apresentam uma grande variedade de propriedades e estruturas químicas. É o efeito combinado de todas as cadeias laterais numa proteína que determina a sua estrutura tridimensional e reatividade química.[39] Os aminoácidos numa cadeia de polipeptídios são unidos por ligações peptídicas. Uma vez unidos na cadeia proteica, cada aminoácido individual é denominado "resíduo", e cada série repetitiva e encadeada de átomos de carbono, nitrogénio e oxigénio é denominada "cadeia principal".[40]
A ligação peptídica tem duas formas de ressonância que contribuem para a formação de uma ligação dupla e inibem a rotação em torno do seu próprio eixo, pelo que os carbonos alfa são aproximadamente coplanares. Os outros dois ângulos diedros na ligação peptídica determinam a forma assumida pela cadeia principal.[41] A extremidade da proteína com um grupo carboxílico livre é denominada C-terminal ou carboxi-terminal, enquanto a extremidade com um grupo livre de amina é denominada N-terminal ou amino-terminal. Os termos "proteína", "polipetídeo" e "peptídeo" são ligeiramente ambíguas e o seu significado pode-se sobrepôr. "Proteína" é geralmente usado para nos referirmos à molécula biológica completa na sua forma terciária estável, enquanto "peptídeo" está geralmente reservado para oligómeros curtos de aminoácidos, aos quais muitas vezes falta uma estrutura tridimensional estável. No entanto, a diferença entre ambos não é bem definida e geralmente corresponde a 20-30 resíduos. "Polipetídeo" pode ser referente a qualquer cadeia linear de aminácidos, independentemente do comprimento, mas onde geralmente não existe uma forma terciária.[42]
Síntese[editar | editar código-fonte]
Biossíntese[editar | editar código-fonte]
As proteínas são produzidas a partir de aminoácidos usando informação codificada nos genes. Cada proteína tem a sua própria sequência de aminoácidos que é especificada pela sequência de nucleótidos do gene que codifica a proteína. O código genético é um grupo de conjuntos com três nucleótidos cada um, denominados codões. Cada uma das combinações de três nucleótidos designa um aminoácido. Por exemplo, AUG (adenina-uracilo-guanina) é o código para a metionina. Uma vez que o ADNcontém quatro nucleótidos, o número total de codões possíveis é de 64. Por este motivo, existe alguma redundância no código genético, havendo alguns aminoácidos que são especificados por mais de um codão.[43] Os genes que são codificados no ADN são inicialmente transcritos para pré-ARN mensageiro (ARNm) por proteínas como aARN-polimerase. A maior parte dos organismos processa em seguida o pré-ARNm, usando várias formas de modificação pós-transcricional, formando assim o ARNm amadurecido, o qual é então usado como molde para a síntese proteica feita pelo ribossoma. Nos procariontes, o ARNm tanto pode ser utilizado assim que é produzido, como ser ligado a um ribossoma depois de se ter afastado do nucleoide. Por outro lado, os eucariontes produzem ARNm no núcleo celular, o qual é depois translocado através do envelope nuclear para o citoplasma, no qual se dá a síntese proteica. A velocidade de síntese proteica é maior nos procariontes do que nos eucariontes, podendo atingir os 20 aminoácidos por segundo.[44]
O processo de síntese de uma proteína a partir de um molde de ARNm é denominado tradução. O ARNm é carregado no ribossoma, no qual são lidos três nucleótidos de cada vez. A leitura é feita fazendo corresponder cada codão com o seu anticodão situado numa molécula de ARN transportador (ARNt), a qual transporta o aminoácido correspondente ao codão por si reconhecido. A enzima aminoacil-tRNA sintetase carrega as moléculas de ARNt com o aminoácido correto. As proteínas são sempre sintetizadas a partir do N-terminal em direção ao C-terminal.[45]
O tamanho de uma proteína sintetizada pode ser medido através do número de aminoácidos e da sua massa molecular total, valor que é geralmente expresso em daltons(Da), sinónimo de unidade de massa atómica. As proteínas das leveduras, por exemplo, têm em média um comprimento de 466 aminoácidos e 53 kDa de massa.[46] As maiores proteínas conhecidas são as titinas, com quase 3000 kDA de massa molecular de 27 000 aminoácidos de comprimento.[47]
Síntese química[editar | editar código-fonte]
As proteínas curtas podem também ser sintetizadas quimicamente através de uma série de métodos denominados síntese de peptídeos, os quais têm por base técnicas de síntese orgânica de elevado rendimento na produção de peptídeos.[48] A síntese química permite a introdução de aminoácidos não naturais nas cadeias de peptídeos.[49] Estes métodos são úteis em laboratórios de bioquímica e biologia celular, embora não se adequem à produção comercial. A síntese química não é eficiente para polipeptídeos maiores do que 300 aminoácidos, e as proteínas sintetizadas podem não assumir imediatamente a sua estrutura terciária nativa. Grande parte dos métodos de síntese química realiza-se a partir do C-terminal em direção ao N-terminal, ao contrário da reação biológica natural.[50]A maior parte das proteínas enovela-se em estruturas tridimensionais distintas. A forma para a qual uma proteína se enovela naturalmente é denominadaconformação nativa.[51] Embora haja muitas proteínas capazes de se enovelar sem assistência, meramente através das propriedades químicas dos seus aminoácidos, há outras que necessitam do auxílio de chaperonas moleculares de modo a se poderem enovelar para a sua conformação nativa.[52] As proteínas podem ter 4 tipos de estruturas dependendo do tipo de aminoácidos, do tamanho da cadeia e da configuração espacial da cadeia polipeptídica: estrutura primária, secundária, terciária e quaternária.[53]
As proteínas não são moléculas completamente rígidas. Para além destes níveis estruturais, as proteínas podem alternar entre várias estruturas enquanto desempenham as suas funções. No contexto destas alterações funcionais, estas estruturas terciárias ou quaternárias são muitas vezes denominadas "conformações", e as transições entre cada uma delas são denominadas "alterações conformacionais". Estas alterações são frequentemente induzidas pela ligação de uma molécula substrato ao sítio ativo de uma enzima – a região física da proteína que participa na catálise química.[54]
As proteínas podem ser divididas informalmente em três classes principais, de acordo com as estruturas terciárias mais comuns: proteínas globulares, proteínas fibrilares e proteínas membranares. Praticamente todas as proteínas globulares são solúveis e grande parte são enzimas. As proteínas fibrilares são muitas vezes estruturais, como é o caso do colagénio, o principal componente do tecido conjuntivo, ou a queratina, a proteína constituinte do cabelo e das unhas. As proteínas membranares atuam muitas vezes como recetores ou proporcionam canais para que as moléculas possam atravessar a membrana celular.[55]
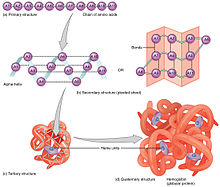
- Estrutura primária
É a sequência linear de aminoácidos ao longo da cadeia polipeptídica da proteína.[56] É o nível estrutural mais simples e mais importante, pois dele deriva todo o arranjo espacial da molécula. É específica para cada proteína, sendo, geralmente, determinada geneticamente. A estrutura primária da proteína resulta numa longa cadeia de aminoácidos, com uma extremidade "amino terminal" e uma extremidade "carboxi terminal".[57]
- Estrutura secundária
É a forma como os aminoácidos se organizam entre si na sequência primária da proteína.[56] Ocorre graças à possibilidade de rotação das ligações entre os carbonos alfa dos aminoácidos e os seus grupos amina e carboxilo. O arranjo secundário de uma cadeia polipeptídica pode ocorrer de forma regular; isso acontece quando os ângulos das ligações entre carbonos alfa e seus ligantes são iguais e se repetem ao longo de um segmento da molécula.[58] A cadeia polipeptídica pode interagir com ela própria através de duas formas principais: pela formação das alfa-hélices e das folhas-beta. Além destas existem estruturas que não são nem hélices nem folhas chamadas laços (loops).[59]
- alfa-hélice: presente na estrutura secundária dos níveis de organização das proteínas. São estruturas cilindricas estabilizadas por pontes de hidrogénio entre aminoácidos. As cadeias laterais dos aminoácidos encontram-se viradas para fora. Existem várias formas de como as hélice alfa podem organizar-se. Na alfa-hélice a espinha dorsal polipeptídica é torcida numa hélice virada à direita.[57]
- folha-beta: padrão estrutural encontrado em várias proteínas, nas quais regiões vizinhas da cadeia polipeptídica associam-se por meio de ligações de hidrogénio, resultando numa estrutura achatada e rígida. Esta é também uma estrutura estável na qual os grupos polares da cadeia polipeptídica associam-se por meio de ligações de hidrogénio um ao outro.[57]
- laços: são secções da sequência que se ligam aos outros dois tipos de estrutura secundária. Em contraste com hélices e folhas, que formam o núcleo da proteína, laços não são estruturas regulares e ficam fora da proteína dobrada.[59]
- Estrutura terciária
A estrutura terciária é a forma como determinada molécula de proteína se organiza no espaço. Corresponde ao movimento, à organização das alfa-hélices, fitas b e voltas no espaço tridimensional, definido por coordenadas atómicas.[60] Resulta do enovelamento das hélices e das folhas pregueadas de uma estrutura secundária e é mantida nessa posição por interações hidrófugas e hidrófilas.[56][61]
- Estrutura quaternária
A estrutura quaternária de uma proteína refere-se à união de várias moléculas proteicas enoveladas num complexo multi-proteico. A interação entre as moléculas é realizada através de ligações não covalentes.[62]
Determinação da estrutura[editar | editar código-fonte]
A descoberta da estrutura terciária de uma proteína, ou da estrutura quaternária dos seus complexos, pode fornecer dados importantes acerca da forma como a proteína realiza a sua função. Entre os métodos mais comuns para determinar a estrutura estão a cristalografia de raios X e a ressonância magnética nuclear de proteínas (RMN), ambas capazes de produzir dados à escala atómica. No entanto, a RMN pode disponibilizar dados a partir dos quais é possível determinar as distâncias entre subconjuntos de pares de átomos, permitindo assim determinar todas as conformações finais possíveis de determinada proteína. A interferometria de dupla polarização é um método analítico que permite medir a conformação e as alterações conformacionais das proteínas em função de interações ou de outros estímulos. O dicroísmo circular é outra técnica laboratorial que permite determinar a composição das proteínas a nível de hélices e folhas beta. A crio-microscopia eletrónica (crio-EM) é usada para produzir informação de baixa resolução sobre complexos proteicos de grande dimensão, entre os quais vírus.[63] Uma variante denominada cristalografia eletrónica é, nalguns casos, capaz de produzir informação de elevada resolução, em particular nos cristais bidimensionais de proteínas membranares.[64]
As estruturas resolvidas são geralmente acrescentadas ao Protein Data Bank (PDB), um recurso disponível gratuitamente que permite consultar os dados estruturais de milhares de proteínas.[65] Conhece-se um número muito maior de sequências genéticas do que estruturas proteicas. Além disso, o conjunto de estruturas resolvidas tende a focar-se naquelas que podem ser facilmente adequadas às condicionantes da cristalografia de raio X. As proteínas globulares, em particular, são as mais fáceis de preparar para a cristalografia de raio X, enquanto as proteínas membranares são difíceis de cristalizar e comparativamente pouco representadas no PDB.[66] As iniciativas de genómica estrutural têm vindo a tentar corrigir esta assimetria através da resolução sistemática das estruturas representativas das principais classes de enovelamento. Para além disso, existem ainda métodos de previsão de estruturas proteicas que tentam fornecer uma estrutura plausível para proteínas cujas estruturas ainda não foram determinadas experimentalmente.[67]
Funções celulares[editar | editar código-fonte]
As proteínas são os principais intervenientes no interior das células, realizando as tarefas determinadas pela informação codificada nos genes.[68] Excetuando determinados tipos de ARN, a maior parte das restantes moléculas biológicas são elementos relativamente inertes nos quais as proteínas atuam. As proteínas são também o principal componente celular; por exemplo, metade do peso de uma célula de Escherichia coli corresponde a proteínas, enquanto outras macromoléculas como o ADN e o ARN correspondem apenas a 3% e 20% do peso, respetivamente. O conjunto de proteínas que podem ser expressas numa determinada célula ou tipo celular é denominadoproteoma.[69]
A principal característica das proteínas, a qual também permite que exerçam um conjunto alargado de funções, é a sua capacidade de ligarem a si outras moléculas, de forma estável e específica. A região da proteína responsável pela agregação de outras moléculas é denominada sítio de ligação, a qual geralmente corresponde a uma depressão na superfície molecular da proteína. Esta capacidade de ligação é mediada pela estrutura terciária da proteína, a qual define a região de ligação, e pelas propriedades químicas das cadeias de aminoácidos laterais. As ligações proteicas podem ser extremamente específicas; por exemplo, a proteína inibidora da ribonucleaseliga-se à angiogenina humana, mas não se liga à sua homóloga anfíbia rampirnase. Há variações químicas extremamente subtis que, por vezes, podem ser o suficiente para eliminar por completo a possibilidade de ligação proteica; por exemplo, o aminoacil-tRNA sintetase específico do aminoácido valina não é capaz de se ligar à cadeia lateral do aminoácido isoleucina, muito similar.[70]
As proteínas podem-se ligar não só a outras proteínas, como também a substratos de pequenas moléculas. Quando as proteínas se ligam especificamente a outras cópias da mesma molécula, são capazes de seoligomerizar para formar fibrilas. Este processo ocorre frequentemente em proteínas estruturais constituídas por monómeros globulares que se associam entre si para formar fibras rígidas. As interações entre proteínas regulam também a atividade enzimática, controlam a progressão ao longo do ciclo celular e permitem a formação de complexos proteicos que desempenham diversas reações no âmbito de uma mesma função biológica. As proteínas também se ligam a membranas celulares. A capacidade de ligarem a si parceiros que induzem alterações conformacionais nas proteínas permite a construção de redes de sinalização celular extremamente complexas.[71] Uma vez que as interações entre proteínas são reversíveis, e dependem da disponibilidade de diferentes grupos de proteínas para formar agregados capazes de desempenhar um conjunto alargado de funções, o estudo das interações entre proteínas específicas é fundamental para compreender aspetos importantes da função celular e, por fim, as propriedades que distinguem diferentes tipos de células.[72]
Enzimática[editar | editar código-fonte]
As proteínas podem atuar na célula enquanto enzimas, as quais são catalisadoras de reações químicas. As enzimas são, regra geral, extremamente específicas e aceleram apenas uma única ou muito poucas reações químicas. As enzimas realizam maior parte das reações que fazem parte do metabolismo e manipulam ADN em vários processos, como a replicação de ADN, reparação de ADN e transcrição genética. Algumas enzimas atuam sobre outras proteínas no sentido de acrescentar ou remover grupos químicos, um processo denominado modificação pós-translacional. São conhecidas cerca de 4000 reações químicas catalisadas por enzimas.[73] A taxa de aceleração proporcionada pela catálise é muitas vezes imensa, podendo chegar a valores na magnitude de 1017 em relação à reação não catalisada, o que faz com que um processo que naturalmente demoraria 78 milhões de anos, com a enzima seja completo em apenas 18 milissegundos.[74] Os compostos químicos que sofrem reações enzimáticas são denominados substratos. Embora as enzimas possam ser constituídas por centenas de aminoácidos, só uma pequena percentagem dos resíduos é que entra em contacto com o substrato e uma percentagem ainda mais pequena – três a quatro resíduos, em média – é que está diretamente envolvida na catálise.[75]
Proteínas estruturais[editar | editar código-fonte]
As proteínas estruturais conferem rigidez a componentes biológicos que, de outra forma, seriam apenas fluidos. A maior parte das proteínas estruturais são proteínas fibrilares; por exemplo, o colagénio e a elastinasão componentes fundamentais do tecido conjuntivo, como a cartilagem, enquanto a queratina está presente em estruturas duras como o cabelo, unhas, penas, cascos e algumas carapaças animais.[76] Algumasproteínas globulares podem também desempenhar funções estruturais; por exemplo, a actina e a tubulina são globulares e solúveis enquanto monómeros, mas são capazes de se polimerizar nas fibras rígidas e longas que formam o citoesqueleto, o qual permite à célula manter a sua forma e tamanho. Outras proteínas que têm funções estruturais são as proteínas motoras como a miosina, cinesina e a dineína, as quais são capazes de gerar força mecânica, como a que contrai os músculos. Estas proteínas são cruciais para a motilidade de organismos unicelulares e dos espermatozoides dos organismos multicelulares que se reproduzem através de reprodução sexuada.[77]
Sinalização celular e proteínas ligantes[editar | editar código-fonte]
Muitas das proteínas estão envolvidas nos processos de sinalização celular e transdução de sinal. Algumas delas, como a insulina, são proteínas extracelulares que transmitem um sinal para outras células em tecidos distantes, a partir da célula onde são sintetizadas. Outras são proteínas membranares que atuam enquanto recetores, cuja principal função é ligar a si uma molécula sinalizadora e induzir uma resposta bioquímica na célula. Muitos dos recetores têm na sua superfície um sítio de ligação e um domínio efetor dentro da célula, o qual pode ter atividade enzimática ou sofrer alterações conformacionais que são detetadas por outras proteínas no interior da célula.[78]
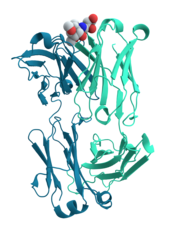
Os anticorpos são componentes proteicos do sistema imunitário adquirido, cuja função principal é ligar a si antigénios ou outras substâncias estranhas ao corpo, marcando-os para serem destruídos. Os anticorpos podem ser segregados para o ambiente extracelular ou ancorados nas membranas de linfócitos B especializados, denominados plasmócitos. Enquanto as enzimas são extremamente limitadas na sua capacidade de ligação, resumindo-se aos substratos necessários para realizar a sua função enzimática, os anticorpos não têm esta restrição, apresentando uma afinidade extremamente elevada.[79]
Muitas das proteínas que transportam ligantes são capazes de ligar a si pequenas biomoléculas, transportando-as para diferentes locais do corpo. Estas proteínas devem ter uma elevada afinidade de ligação nos casos em que o seu ligante esteja presente em elevada concentração, mas serem também capazes de libertar o ligante nos casos em que a sua concentração nos tecidos-alvo seja diminuta. O exemplo canónico de uma proteína ligante é a hemoglobina, a qual transporta o oxigénio dos pulmões para os restantes órgãos e tecidos em todos os vertebrados e tem homólogos em todos os reinos.[80] As proteínas transmembranares atuam também enquanto proteínas transportadoras de ligantes, capazes de alterar a permeabilidade da membrana celular em relação a pequenas moléculas e a iões. A própria membrana tem um núcleo hidrófugo, através do qual as moléculaspolarizadas ou carregadas eletrónicamente não são capazes de se difundir. As proteínas membranares possuem canais internos que permitem a este tipo de moléculas entrar e sair da célula. Muitas proteínas com canais iónicos são especializadas no sentido de selecionar apenas um ião em particular; por exemplo, os canais de potássio e sódio muitas vezes aceitam apenas um dos dois iões.[81]
Doenças proteicas[editar | editar código-fonte]
A acumulação de proteínas mal enoveladas pode causar doenças amiloides, um grupo de várias doenças comuns, entre as quais a doença de Alzheimer, doença de Parkinson e doença de Huntington. O risco destas doenças aumenta significativamente com a idade. À medida que o ser humano envelhece, o equilíbrio da síntese, enovelamento e degradação das proteínas vai sofrendo distúrbios, o que provoca a acumulação de proteínas mal enoveladas em agregados. No entanto, as doenças causadas pela agregação de proteínas mal enoveladas não são exclusivas do sistema nervoso central e podem-se manifestar em tecidos periféricos, como no caso da diabetes mellitus tipo 2, cataratas hereditárias, algumas formas de aterosclerose, distúrbios relacionados com a hemodiálise e amiloidose, entre outras. Os genes e produtos proteicos envolvidos nestas doenças denominam-se amiloidogénicos e todas estas doenças têm em comum a expressão de uma proteína fora do seu contexto normal. Em todas estas doenças, a agregação de proteínas pode ser causada por mero acaso, por hiperfosforilação proteica, por mutações que tornam a proteína instável ou ainda pelo aumento desregulado ou patológico da concentração de algumas destas proteínas entre as células. Estes desiquilíbrios na concentração podem ser causados por mutações dos genes amiloidogénicos, alterações na sequência de aminoácidos da proteína ou por deficiências noproteassoma.[82]
Métodos de estudo[editar | editar código-fonte]
As proteínas são uma das moléculas biológicas mais intensivamente estudadas, quer in vitro, in vivo ou in silico. O estudo in vitro de proteínas purificadas em ambiente controlado é útil na aprendizagem da forma como as proteínas desempenham as suas funções; por exemplo, o estudo da cinética enzimática explora o mecanismo químico da atividade catalítica de uma enzima e a sua afinidade relativa em relação às possíveis moléculas substrato. Por outro lado, as experiências in vivo podem fornecer dados sobre o papel fisiológico da proteína no contexto de uma célula ou de um organismo. O estudo in silico recorre a métudos computacionais para estudar proteínas.
Purificação de proteínas[editar | editar código-fonte]
Antes de se poder efetuar uma análise in vitro, a proteína deve ser purificada dos restantes componentes celulares. Este processo de purficação geralmente tem início com a citólise da célula, através da qual a membrana celular é rompida e o conteúdo interno libertado para uma solução denominada lisado bruto. A mistura daí resultante pode ser purificada através de ultracentrifugação, a qual fracciona os vários componentes celulares em frações que contêm proteínas solúveis, proteínas e lípidos membranares, organelas celulares e ácidos nucleicos. As proteínas deste lisado são então concetradas através deprecipitação, feita através do método de relargagem. São depois usados vários tipos de cromatografia para isolar a proteína ou as proteínas pretendidas, de acordo com propriedades como o peso molecular ou afinidade de ligação.[83] O nível de purificação pode ser monitorizado através de vários tipos de eletroforese em gel, quando são conhecidos o peso molecular e o ponto isoelétrico, através de espectroscopia, quando a proteína possui características espectroscópicas distintas, ou através de análise enzimática, quando a enzima tem atividade enzimática. As proteínas podem ainda ser isoladas de acordo com a sua carga através de focalização isoelétrica.[84]
No caso das proteínas naturais, podem ser necessárias mais etapas no processo de purificação de forma a obter proteínas suficientemente puras para serem usadas em laboratório. Para simplificar este processo, recorre-se muitas vezes a engenharia genética para acrescentar às proteínas características químicas que as tornem mais fáceis de serem purificadas sem, no entanto, afetar a sua estrutura ou atividade. Neste caso, a um dos terminais da proteína é acrescentada uma etiqueta constituída por uma sequência de aminoácidos específica, geralmente uma série de resíduos de histidina (etiqueta de poli-histidina). Desta forma, quando o lisado é passado sobre uma coluna de cromatografia contendo níquel, os resíduos de histidina ligam-se com o níquel e agarram-se à coluna, enquanto os componentes do lisado sem a etiqueta passam sem entraves. Têm vindo a ser desenvolvidas diversas etiquetas de modo a auxiliar os investigadores na purificação de proteínas a partir de misturas complexas.[85]
Localização celular[editar | editar código-fonte]
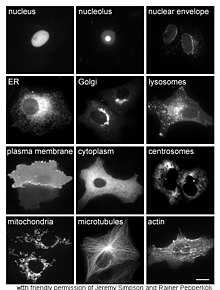
O estudo de proteínas in vivo dedica-se às questões relacionadas com a síntese e localização de proteínas no interior de células. Embora muitas das proteínas intrecelulares sejam sintetizadas no citoplasma e as proteínas segregadas sejam sintetizadas no retículo endoplasmático, o processo específico de como as proteínas se orientam para organelas ou estruturas celulares específicas é em muitas situações pouco claro. Uma das técnicas para avaliar a localização celular recorre a engenharia genética para expressar numa célula uma proteína de fusão, a qual é constituída pela proteína em estudo ligada a um gene repórter, como por exemplo a proteína verde fluorescente.[86] A posição da proteína de fusão na célula pode então ser facilmente visualizada através de microscopia.[87]
Outros métodos para obtenção da localização celular de proteínas requerem o uso de marcadores compartimentais conhecidos para diversas regiões celulares. Com o uso de versões destes marcadores etiquetadas com fluorescência, torna-se mais simples a identificação e localização da proteína pretendida.[88]
A técnica padrão para localização celular é a microscopia imunoeletrónica. Esta técnica usa um anticorpo para a proteína pretendida, a par de técnicas clássicas de microscopia eletrónica. A amostra é preparada para uma análise microscópica padrão, sendo depois tratada com um anticorpo dessa proteína que é conjugado com um material eletro-denso, geralmente ouro.[89] Através de mutagénese sítio-dirigida, os investigadores podem alterar a sequência proteica, alterando dessa forma a sua estrutura, localização celular e suscetibilidade à regulação. Esta técnica permite ainda a incorporação nas proteínas de aminoácidos não naturaus, usando ARNt modificado,[90] podendo ainda permitir a conceção de novas proteínas com novas proriedades.[91]O conjunto total de proteínas presentes numa célula em determinado momento é denominado proteoma, e o estudo em grande escala destes conjuntos define o campo daproteómica, assim denominado em analogia ao campo relacionado da genómica. Entre as principais técnicas da proteómica estão a eletroforese bidimensional,[92] a qual permite a separação de um vasto número de proteínas, a espectrometria de massa,[93] a qual permite a rápida identificação de proteínas e sequenciação de peptídeos, o microarranjo de proteínas,[94] que permite a deteção dos níveis relativos do grande número de proteínas presentes na célula, e o sistema de duplo híbrido, que permite a exploração sistemática de interações proteína-proteína.[95] O conjunto total e biologicamente possível destas interações é denominado interactoma.[96] O esforço sistemático para determinar as estruturas de proteínas e de todos os enovelamentos possíveis é denominado genómica estrutural.[97]
Previsão e simulação da estrutura[editar | editar código-fonte]
Complementar ao campo da genómica estrutural, a previsão de estruturas das proteínas procura desenvolver métodos eficientes de fornecer modelos plausíveis para proteínas cujas estruturas não foram ainda determinadas experimentalmente.[98] O mais bem-sucedido método de previsão estrutural, denominado modelação por homologia, assenta na existência de uma estrutura-modelo com uma sequência semelhante à proteína a ser modelada. O objetivo da genómica estrutural é fornecer uma representatividade suficiente de estruturas resolvidas que sirva de modelo a todas as restantes.[99]
Os processos de enovelamento e ligação proteica podem ser simulados usando técnicas como a dinâmica molecular ou o método de Monte Carlo, os quais têm vindo cada vez mais a tirar partido da computação distribuída, como o projeto Folding@home.[100] O enovelamento de pequenos domínios proteicos de alfa-hélice, como a proteína acessória do VIH, tem vindo a ser simulado com sucesso in silico.[101] Os métodos híbridos que combinam dinâmica molecular com cálculo de mecânica quântica têm permitido a exploração dos estados eletrónicos das rodopsinas.[102]
História e etimologia[editar | editar código-fonte]
As proteínas foram pela primeira vez descritas pelo químico holandês Gerardus Johannes Mulder e assim batizadas pelo químico sueco Jöns Jacob Berzelius em 1838. Mulder levou a cabo análises elementares de proteínas vulgares e constatou que praticamente todas as proteínas apresentavam a mesma fórmula empírica – C400H620N100O120P1S1. Ainda que erradamente, concluiu que as proteínas deveriam ser constituídas por um único tipo de molécula de grande dimensão.[103] O termo "proteína" para descrever estas moléculas foi proposto pelo sócio de Mulder, Berzelius. Proteína deriva da palavra grega πρωτεῖος (proteios), a qual significa "na liderança" ou "a que está à frente".[104] Mulder prossegui a investigação, identificando produtos da degradação proteica, como o aminoácido leucina, para o qual determinou o peso molecular quase preciso de 131 Da.[105]
Os cientistas pioneiros no campo da nutrição, como o alemão Carl von Voit, acreditavam que a proteína era o mais importante nutriente na manutenção da estrutura corporal, uma vez que existia a crença generalizada de que seria a carne tinha origem na própria carne.[106] Karl Heinrich Ritthausen alargou o campo das proteínas conhecidas com a identificação do ácido glutâmico. Thomas Burr Osborne compilou em 1909 uma revisão detalhada de todas as proteínas vegetais e, no mesmo ano e em conjunto com Lafayette Mendel, determinou os aminoácidos essenciais à sobrevivência de ratos de laboratório aplicando a lei de Liebig. A compreensão das proteínas enquanto polipetídeos foi proporcionada por Franz Hofmeister e Hermann Emil Fischer. O papel central das proteínas enquanto enzimas nos organismos vivos foi determinado em 1926, quando James Batcheller Sumner demonstrou que a urease era de facto uma proteína.[107]
A dificuldade em purificar proteínas em grande quantidade dificultou imenso a investigação dos primeiros bioquímicos. Assim, a investigação inicial focou-se sobretudo em proteínas que podiam ser facilmente purificadas em quantidade, como as do sangue, da clara de ovo, diversas toxinas e enzimas digestivas obtidas em matadouros.[108]Atribiu-se a Linus Pauling a primeira previsão bem-sucedida de de estruturas secundárias de proteínas com base nas ligações de hidrogénio, uma ideia que já tinha sido proposta em 1933 por William Astbury.[109] Posteriormente, a investigação de Walter Kauzmann sobre a desnaturação, baseada em parte nos estudos anteriores de Kaj Ulrik Linderstrøm-Lang, veio a contribuir para a compreensão do enovelamento de proteínas e das estruturas mediadas por interações hidrófugas.[110][111][112] A primeira proteína a ser sequenciada foi a insulina, por Frederick Sanger em 1949. Sanger determinou corretamente a sequência de aminoácidos da proteína, demonstrando de forma conclusiva que as proteínas eram constituídas por polímeros lineares de aminoácidos, em vez de cadeias ramificadas ou coloides.[113]
As primeiras estruturas proteicas a serem resolvidas foram as da hemoglobina e da mioglobina, por Max Perutz e John Kendrew, respetivamente, em 1958.[114][115] Nas décadas posteriores, a crio-microscopia eletrónica de grandes conjuntos macromoleculares e a previsão computacional de estruturas proteicas de pequenos domínios foram métodos que permitiram a investigação de proteínas à escala atómica.[116][117] No início de 2014, estavam registadas no Protein Data Bank aproximadamente 90 000 estruturas proteicas com resolução atómica.[118]
Referências
- ↑ Hermann 1995
- ↑ Voet 2004
- ↑ Brosnan 2003
- ↑ USDA 2009, p. 192
- ↑ Genton 2010, pp. 413-423
- ↑ a b c Evert 2009
- ↑ Genton 2010, pp. 413-423
- ↑ Hermann 1995
- ↑ Food and Nutrition Board 2005
- ↑ Food and Nutrition Board 2005
- ↑ Hermann 1995
- ↑ IOC 2003
- ↑ IOC 2003
- ↑ Vernon 1994
- ↑ Steinke 1992, pp. 91–100
- ↑ Vernon 1994
- ↑ Hermann 1995
- ↑ FAO 1985
- ↑ Latham 1997
- ↑ Vernon 1994
- ↑ FAO 1985
- ↑ FAO 1985
- ↑ a b c d Bilsborough 2006, pp. 129–152
- ↑ a b c Lemon 2000, pp. 513-521
- ↑ a b c d Tarnopolsky 1992, pp. 1986-95
- ↑ Genton 2010, pp. 413-423
- ↑ Lemon 2008, pp. S39–61
- ↑ 2007
- ↑ a b EFSA 2012
- ↑ Institute of Medicine 2005
- ↑ a b Food and Nutrition Board 2005
- ↑ Ten Have 2007
- ↑ Elliott 2006, pp. 79-87
- ↑ Latham 1997
- ↑ USDA 2009, p. 192
- ↑ Otten 2006
- ↑ Zeratsky 2015
- ↑ Nelson 2005
- ↑ Gutteridge 2005
- ↑ Murray 2006, p. 19
- ↑ Murray 2006, p. 31
- ↑ Lodish 2004
- ↑ vanHolde 1996
- ↑ Pain 2000
- ↑ Van Holde 1996, pp. 1002-1042
- ↑ Lodish 2004
- ↑ Fulton 1991
- ↑ Bruckdorfer 2004
- ↑ Schwarzer 2005
- ↑ Kent 2009
- ↑ Murray 2006, pp. 30-34
- ↑ Murray 2006, p. 37
- ↑ Alphey 1997, p. 179
- ↑ Van Holde 1996, pp. 368-375
- ↑ Van Holde 1996, pp. 165-185
- ↑ a b c Karp 2008, pp. 49-64
- ↑ a b c Sharma 2009, pp. 2-6
- ↑ Berg 2002
- ↑ a b Setubal 1997, pp. 252-253
- ↑ IUPAC 1997
- ↑ biology-online.org s. d.
- ↑ biology-online.org s. d. b
- ↑ Branden 1999, pp. 340-341
- ↑ Gonen 2005
- ↑ Stanley 2008
- ↑ Walian 2004
- ↑ Sleator 2012
- ↑ Lodish 2004
- ↑ Voet 2004
- ↑ Sankaranarayanan 2001
- ↑ Van Holde 1996, pp. 830–49
- ↑ Samarin 2009
- ↑ Bairoch 2000
- ↑ Radzicka 1995
- ↑ EBI 2010
- ↑ Van Holde 1996, pp. 178-81
- ↑ Van Holde 1996, pp. 258-264, 272
- ↑ Branden 1999, pp. 251–81
- ↑ Van Holde 1996, pp. 247–250
- ↑ Van Holde 1996, pp. 220–229
- ↑ Branden 1999, pp. 232-34
- ↑ Reynaud 2010, p. 28
- ↑ Murray 2006, pp. 21-24
- ↑ Hey 2008
- ↑ Terpe 2003
- ↑ Stepanenko 2008
- ↑ Yuste 2005
- ↑ Margolin 2000
- ↑ Mayhew 2008
- ↑ Hohsaka 2002
- ↑ Cedrone 2000
- ↑ Gorg 2008
- ↑ Conrotto 2008
- ↑ Joos 2009
- ↑ Koeg 2007
- ↑ Plewczynski 2009
- ↑ Zhang 2003
- ↑ Zhang 2008
- ↑ Xiang 2006
- ↑ Scheraga 2007
- ↑ Herges 2005
- ↑ Hoffman 2006
- ↑ Perrett 2007
- ↑ Reynolds 2003
- ↑ Perrett 2007
- ↑ Bischoff 1860
- ↑ Sumner 1926
- ↑ Perrett 2007
- ↑ Pauling 1951
- ↑ Kalman 1955
- ↑ Kauzmann 1956
- ↑ Kauzmann 1959
- ↑ Sanger 1949
- ↑ Muirhead 1963
- ↑ Kendrew 1958
- ↑ Zhou 2008
- ↑ keskin 2008
- ↑ RCSB s. d.
Bibliografia[editar | editar código-fonte]
- Alphey, Luke (1997). «17: Protein Structure Prediction». DNA Sequencing. From Experimental Methods to Bioinformatics (em inglês) (New York: Springer). p. 179. ISBN 0-387-91509-5.
- Bilsborough, Shane; Neil Mann. (2006). "A Review of Issues of Dietary Protein Intake in Humans" (16).
- Bischoff, TLW; Voit, C (1860). Die Gesetze der Ernaehrung des Pflanzenfressers durch neue Untersuchungen festgestellt (em German) (Leipzig, Heidelberg [s.n.]).
- Brosnan, J (June 2003). «Interorgan amino acid transport and its regulation». Journal of Nutrition[S.l.: s.n.] 133 (6 Suppl 1): 2068S–72S. PMID 12771367.
- Bruckdorfer, T; Marder, O; Albericio, F (2004). «From production of peptides in milligram amounts for research to multi-tons quantities for drugs of the future». Current Pharmaceutical Biotechnology [S.l.: s.n.] 5 (1): 29–43. doi:10.2174/1389201043489620. PMID 14965208.
- Cedrone, F; Ménez, A; Quéméneur, E (2000). «Tailoring new enzyme functions by rational redesign».Current Opinion in Structural Biology [S.l.: s.n.] 10 (4): 405–10. doi:10.1016/S0959-440X(00)00106-8. PMID 10981626.
- Conrotto, P; Souchelnytskyi, S (2008). «Proteomic approaches in biological and medical sciences: principles and applications». Experimental Oncology [S.l.: s.n.] 30 (3): 171–80. PMID 18806738.
- Copland JA; Sheffield-Moore M; Koldzic-Zivanovic N; Gentry S; Lamprou G; Tzortzatou-Stathopoulou F,;Zoumpourlis V, Urban RJ; Vlahopoulos SA (2009). «Sex steroid receptors in skeletal differentiation and epithelial neoplasia: is tissue-specific intervention possible?». BioEssays: news and reviews in molecular, cellular and developmental biology [S.l.: s.n.] 31 (6): 629–41.doi:10.1002/bies.200800138. PMID 19382224.
- Bairoch, A. (2000). "The ENZYME database in 2000". Nucleic Acids Research 28 (1): 304–305.DOI:10.1093/nar/28.1.304. PMID 10592255.
- Fernández, A; Scott, R (2003). «Dehydron: a structurally encoded signal for protein interaction».Biophysical Journal [S.l.: s.n.] 85 (3): 1914–28. Bibcode:2003BpJ....85.1914F. doi:10.1016/S0006-3495(03)74619-0. PMC 1303363. PMID 12944304.
- Fulton, A; Isaacs, W (1991). «Titin, a huge, elastic sarcomeric protein with a probable role in morphogenesis». BioEssays [S.l.: s.n.] 13 (4): 157–61. doi:10.1002/bies.950130403.PMID 1859393.
- Gonen T; Cheng Y; Sliz P; Hiroaki Y; Fujiyoshi Y; Harrison SC; Walz T (2005). «Lipid-protein interactions in double-layered two-dimensional AQP0 crystals». Nature [S.l.: s.n.] 438 (7068): 633–38. Bibcode:2005Natur.438..633G. doi:10.1038/nature04321. PMC 1350984.PMID 16319884.
- Görg A; Weiss W; Dunn MJ (2004). «Current two-dimensional electrophoresis technology for proteomics». Proteomics [S.l.: s.n.] 4 (12): 3665–85. doi:10.1002/pmic.200401031.PMID 15543535.
- Gutteridge, A; Thornton, JM (2005). «Understanding nature's catalytic toolkit». Trends in Biochemical Sciences [S.l.: s.n.] 30 (11): 622–29. doi:10.1016/j.tibs.2005.09.006. PMID 16214343.
- Herges, T; Wenzel, W (2005). «In silico folding of a three helix protein and characterization of its free-energy landscape in an all-atom force field». Physical Review Letters [S.l.: s.n.] 94 (1): 018101.Bibcode:2005PhRvL..94a8101H. doi:10.1103/PhysRevLett.94.018101. PMID 15698135.
- Hey J; Posch A; Cohen A; Liu N; Harbers A (2008). «Fractionation of complex protein mixtures by liquid-phase isoelectric focusing». Methods in Molecular Biology. Methods in Molecular Biology [S.l.: s.n.] 424: 225–39. doi:10.1007/978-1-60327-064-9_19. ISBN 978-1-58829-722-8.PMID 18369866.
- Hoffmann M; Wanko M; Strodel P; König PH; Frauenheim T; Schulten K; Thiel W; Tajkhorshid E; Elstner M (2006). «Color tuning in rhodopsins: the mechanism for the spectral shift between bacteriorhodopsin and sensory rhodopsin II». Journal of the American Chemical Society [S.l.: s.n.] 128(33): 10808–18. doi:10.1021/ja062082i. PMID 16910676.
- Hohsaka, T; Sisido, M (2002). «Incorporation of non-natural amino acids into proteins». Current Opinion in Chemical Biology [S.l.: s.n.] 6 (6): 809–15. doi:10.1016/S1367-5931(02)00376-9.PMID 12470735.
- IUPAC (1997). A. D. McNaught, A. Wilkinson., : . Compendium of Chemical Terminology 2ª ed. (Oxford: Blackwell Scientific Publications). doi:10.1351/goldbook. Verifique
|doi=(Ajuda). ISBN 0-9678550-9-8. Verifique|isbn=(Ajuda).
- Joos T, Bachmann J (2009). «Protein microarrays: potentials and limitations». Frontiers in Bioscience [S.l.: s.n.] 14 (14): 4376–85. doi:10.2741/3534. PMID 19273356.
- Kalman SM, Linderstrom-Lang K, Ottesen M, Richards FM (1955). «Degradation of ribonuclease by subtilisin». Biochimica et Biophysica Acta [S.l.: s.n.] 16 (2): 297–99. doi:10.1016/0006-3002(55)90224-9. PMID 14363272.
- Karp, Gerald (2008). Cell and Molecular Biology. Concepts and Experiments (em inglês) 5ª ed. (Nova Jérsia: John Wiley). ISBN 978-0-470-04217-5.
- Kauzmann W (1956). «Structural factors in protein denaturation». Journal of Cellular Physiology. Supplement [S.l.: s.n.] 47 (Suppl 1): 113–31. doi:10.1002/jcp.1030470410. PMID 13332017.
- Kauzmann, W (1959). «Some factors in the interpretation of protein denaturation». Advances in Protein Chemistry. Advances in Protein Chemistry [S.l.: s.n.] 14: 1–63. doi:10.1016/S0065-3233(08)60608-7. ISBN 978-0-12-034214-3. PMID 14404936.
- Kendrew J; Bodo G; Dintzis H; Parrish R; Wyckoff H; Phillips D (1958). «A three-dimensional model of the myoglobin molecule obtained by X-ray analysis». Nature [S.l.: s.n.] 181 (4610): 662–66.Bibcode:1958Natur.181..662K. doi:10.1038/181662a0. PMID 13517261.
- Kent, SB (2009). «Total chemical synthesis of proteins». Chemical Society Reviews [S.l.: s.n.] 38 (2): 338–51. doi:10.1039/b700141j. PMID 19169452.
- Keskin O; Tuncbag N; Gursoy A (2008). «Characterization and prediction of protein interfaces to infer protein-protein interaction networks». Current Pharmaceutical Biotechnology [S.l.: s.n.] 9 (2): 67–76.doi:10.2174/138920108783955191. PMID 18393863.
- Koegl, M; Uetz, P (2007). «Improving yeast two-hybrid screening systems». Briefings in Functional Genomics & Proteomics [S.l.: s.n.] 6 (4): 302–12. doi:10.1093/bfgp/elm035. PMID 18218650.
- Kuhlman B; Dantas G; Ireton GC; Varani G; Stoddard BL; Baker D (2003). «Design of a novel globular protein fold with atomic-level accuracy». Science [S.l.: s.n.] 302 (5649): 1364–68.Bibcode:2003Sci...302.1364K. doi:10.1126/science.1089427. PMID 14631033.
- Lemon, Peter. (2000). "Beyond the Zone: Protein Needs of Active Individuals" 19.DOI:10.1080/07315724.2000.10718974. PMID 11023001.
- Lemon, PW. (Junho 2008). "Do athletes need more dietary protein and amino acids?". Int J Sport Nutr.5 Suppl. PMID 7550257.</ref>
- Lodish H; Berk A; Matsudaira P; Kaiser CA; Krieger M; Scott MP; Zipurksy SL; Darnell J (2004).Molecular Cell Biology 5th ed. (New York, New York: WH Freeman and Company).
- Margolin, W (2000). «Green fluorescent protein as a reporter for macromolecular localization in bacterial cells». Methods (San Diego, Calif.) [S.l.: s.n.] 20 (1): 62–72.doi:10.1006/meth.1999.0906. PMID 10610805.
- Mayhew, TM; Lucocq, JM (2008). «Developments in cell biology for quantitative immunoelectron microscopy based on thin sections: a review». Histochemistry and Cell Biology [S.l.: s.n.] 130 (2): 299–313. doi:10.1007/s00418-008-0451-6. PMC 2491712. PMID 18553098.
- Muirhead, H; Perutz, M (1963). «Structure of hemoglobin. A three-dimensional fourier synthesis of reduced human hemoglobin at 5.5 Å resolution». Nature [S.l.: s.n.] 199 (4894): 633–38.Bibcode:1963Natur.199..633M. doi:10.1038/199633a0. PMID 14074546.
- Nelson, DL; Cox, MM (2005). Lehninger's Principles of Biochemistry 4th ed. (New York, New York: W. H. Freeman and Company).
- Dobson, CM (2000). «The nature and significance of protein folding». In: Pain RH (ed.). Mechanisms of Protein Folding (Oxford, Oxfordshire: Oxford University Press). pp. 1–28. ISBN 0-19-963789-X.
- Pauling L; Corey RB; Branson HR (1951). «The structure of proteins: two hydrogen-bonded helical configurations of the polypeptide chain» (PDF). Proceedings of the National Academy of Sciences U.S.A. [S.l.: s.n.] 37 (5): 235–40. Bibcode:1951PNAS...37..235P. doi:10.1073/pnas.37.5.235.PMC 1063348. PMID 14834145.
- Perrett, D (2007). «From 'protein' to the beginnings of clinical proteomics». Proteomics – Clinical Applications [S.l.: s.n.] 1 (8): 720–38. doi:10.1002/prca.200700525. PMID 21136729.
- Plewczyński, D; Ginalski, K (2009). «The interactome: predicting the protein–protein interactions in cells». Cellular & Molecular Biology Letters [S.l.: s.n.] 14 (1): 1–22. doi:10.2478/s11658-008-0024-7.PMID 18839074.
- Radzicka, A; Wolfenden, R (1995). «A proficient enzyme». Science [S.l.: s.n.] 267 (5194): 90–93.Bibcode:1995Sci...267...90R. doi:10.1126/science.7809611. PMID 7809611.
- Reynaud, E. . (2010). "Protein Misfolding and Degenerative Diseases". Nature Education 3 (9).
- Reynolds, JA; Tanford, C (2003). Nature's Robots: A History of Proteins (Oxford Paperbacks) (New York, New York: Oxford University Press). p. 15. ISBN 0-19-860694-X.
- Ritchie, DW (2008). «Recent progress and future directions in protein–protein docking». Current Protein and Peptide Science [S.l.: s.n.] 9 (1): 1–15. doi:10.2174/138920308783565741.PMID 18336319.
- Rüdiger H; Siebert HC; Solís D; Jiménez-Barbero J; Romero A; von der Lieth CW; Diaz-Mariño T; Gabius HJ (2000). «Medicinal chemistry based on the sugar code: fundamentals of lectinology and experimental strategies with lectins as targets». Current Medicinal Chemistry [S.l.: s.n.] 7 (4): 389–416.doi:10.2174/0929867003375164. PMID 10702616.
- Samarin, S; Nusrat, A (2009). «Regulation of epithelial apical junctional complex by Rho family GTPases». Frontiers in bioscience: a journal and virtual library [S.l.: s.n.] 14 (14): 1129–42.doi:10.2741/3298. PMID 19273120.
- Sanger, F (1949). «The terminal peptides of insulin». Biochemical Journal [S.l.: s.n.] 45 (5): 563–74.PMC 1275055. PMID 15396627.
- Sankaranarayanan, R; Moras, D (2001). «The fidelity of the translation of the genetic code». Acta Biochimica Polonica [S.l.: s.n.] 48 (2): 323–35. PMID 11732604.
- Scheraga, HA; Khalili M, Liwo A (2007). «Protein-folding dynamics: overview of molecular simulation techniques». Annual Review of Physical Chemistry [S.l.: s.n.] 58: 57–83.Bibcode:2007ARPC...58...57S. doi:10.1146/annurev.physchem.58.032806.104614.PMID 17034338.
- Schwarzer, D; Cole, P (2005). «Protein semisynthesis and expressed protein ligation: chasing a protein's tail». Current Opinion in Chemical Biology [S.l.: s.n.] 9 (6): 561–69.doi:10.1016/j.cbpa.2005.09.018. PMID 16226484.
- Setubal, João; Meidanis, João (1997). «8: Molecular Structure Predition». Introduction to Computational Molecular Biology. edição= (Boston: PWS Publishing Company). ISBN 0-534-95262-3.
- Sharma, Kal Renganathan (2009). Bioinformatics (Nova Iorque: McGraw-Hill). ISBN 978-0-07-159306-9.
- Sleator, RD. (2012). «Prediction of protein functions». Methods in Molecular Biology. Methods in Molecular Biology [S.l.: s.n.] 815: 15–24. doi:10.1007/978-1-61779-424-7_2. ISBN 978-1-61779-423-0. PMID 22130980.
- Standley DM; Kinjo AR; Kinoshita K; Nakamura H (2008). «Protein structure databases with new web services for structural biology and biomedical research». Briefings in Bioinformatics [S.l.: s.n.] 9 (4): 276–85. doi:10.1093/bib/bbn015. PMID 18430752.
- Stepanenko OV; Verkhusha VV; Kuznetsova IM; Uversky VN; Turoverov KK (2008). «Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes».Current Protein & Peptide Science [S.l.: s.n.] 9 (4): 338–69. doi:10.2174/138920308785132668.PMC 2904242. PMID 18691124.
- Sumner, JB (1926). «The isolation and crystallization of the enzyme urease. Preliminary paper»(PDF). Journal of Biological Chemistry [S.l.: s.n.] 69 (2): 435–41.
- Tarnopolsky, MA; Atkinson SA, MacDougall JD, Chesley A, Phillips S, Schwarcz HP. (1992). "Evaluation of protein requirements for trained strength athletes". Journal of Applied Physiology 73 (5).PMID 1474076.
- Terpe, K (2003). «Overview of tag protein fusions: from molecular and biochemical fundamentals to commercial systems». Applied Microbiology and Biotechnology [S.l.: s.n.] 60 (5): 523–33.doi:10.1007/s00253-002-1158-6. PMID 12536251.
- EBI (20 de janeiro de 2010). «The Catalytic Site Atlas at The European Bioinformatics Institute». Ebi.ac.uk. Consultado em 16 de janeiro de 2011.
- Walian P; Cross TA; Jap BK (2004). «Structural genomics of membrane proteins». Genome Biology[S.l.: s.n.] 5 (4): 215. doi:10.1186/gb-2004-5-4-215. PMC 395774. PMID 15059248.
- Walker, JH; Wilson, K (2000). Principles and Techniques of Practical Biochemistry (Cambridge, UK: Cambridge University Press). pp. 287–89. ISBN 0-521-65873-X.
- Xiang, Z (2006). «Advances in homology protein structure modeling». Current Protein and Peptide Science [S.l.: s.n.] 7 (3): 217–27. doi:10.2174/138920306777452312. PMC 1839925.PMID 16787261.
- Yuste, R (2005). «Fluorescence microscopy today». Nature Methods [S.l.: s.n.] 2 (12): 902–904.doi:10.1038/nmeth1205-902. PMID 16299474.
- Zagrovic B; Snow CD;, Shirts MR; Pande VS (2002). «Simulation of folding of a small alpha-helical protein in atomistic detail using worldwide-distributed computing». Journal of Molecular Biology [S.l.: s.n.] 323 (5): 927–37. doi:10.1016/S0022-2836(02)00997-X. PMID 12417204.
- Zhang, C; Kim, SH (2003). «Overview of structural genomics: from structure to function». Current Opinion in Chemical Biology [S.l.: s.n.] 7 (1): 28–32. doi:10.1016/S1367-5931(02)00015-7.PMID 12547423.
- Zhang, Y; Skolnick, J (2005). «The protein structure prediction problem could be solved using the current PDB library». Proceedings of the National Academy of Sciences U.S.A. [S.l.: s.n.] 102 (4): 1029–34. Bibcode:2005PNAS..102.1029Z. doi:10.1073/pnas.0407152101. PMC 545829.PMID 15653774.
- Zhang, Y (2008). «Progress and challenges in protein structure prediction». Current Opinion in Structural Biology [S.l.: s.n.] 18 (3): 342–48. doi:10.1016/j.sbi.2008.02.004. PMC 2680823.PMID 18436442.
- Zhou, ZH (2008). «Towards atomic resolution structural determination by single-particle cryo-electron microscopy». Current Opinion in Structural Biology [S.l.: s.n.] 18 (2): 218–28.doi:10.1016/j.sbi.2008.03.004. PMC 2714865. PMID 18403197.
- Branden, C; Tooze, J (1999). Introduction to Protein Structure (Nova Iorque: Garland Pub). ISBN 0-8153-2305-0.
- Murray, RF; Harper HW; Granner DK; Mayes PA; Rodwell VW (2006). Harper's Illustrated Biochemistry(Nova Iorque: Lange Medical Books/McGraw-Hill). ISBN 0-07-146197-3.
- Van Holde, KE; Mathews, CK (1996). Biochemistry (Menlo Park, California: Benjamin/Cummings Pub. Co., Inc). ISBN 0-8053-3931-0.
- Voet, D; Voet JG (2004). Biochemistry 1 3ª ed. (Hoboken: Wiley). ISBN 978-0470917459. </ref>
- Berg, JM; Tymoczko JL, Stryer L (2002). Biochemistry 5 ed. (Nova Iorque: W H Freeman).
- Lubert Stryer, Jeremy Mark Berg, John L. Tymoczko (trad. Serge Weinman), Biochimie, Flammarion, « Médecine-Sciences », Paris, 2003, 5ª éd. ISBN 2-257-17116-0.
- Carl-Ivar Brändén, John Tooze (trad. Bernard Lubochinsky, préf. Joël Janin), Introduction à la structure des protéines, De Boeck Université, Bruxelles, 1996 ISBN 2-804-12109-7.
- Rodríguez, Faride. La estructura de las proteínas
- Lehninger, Albert L. Princípios da bioquímica
- Hermann, Janice R.. (1995). "Protein and the Body". Oklahoma Cooperative Extension Service, Division of Agricultural Sciences and Natural Resources • Oklahoma State University: T–3163–1 – T–3163–4.
- Genton, Laurence; Melzer, Katarina; Pichard, Claude. (2010). "Energy and macronutrient requirements for physical fitness in exercising subjects". Clinical Nutrition 29 (4): 413–423.DOI:10.1016/j.clnu.2010.02.002. PMID 20189694.
- Food and Nutrition Board (2005). A Report of the Panel on Macronutrients, Subcommittees on Upper Reference Levels of Nutrients and Interpretation and Uses of Dietary Reference Intakes, and the Standing Committee on the Scientific Evaluation of Dietary Reference Intakes. Dietary Reference Intakes for Energy, Carbohydrate, Fiber, Fat, Fatty Acids, Cholesterol, Protein, and Amino Acids (Macronutrients) THE NATIONAL ACADEMIES PRESS, Washington, D.C. [S.l.] ISBN 0-309-08537-3.
- IOC, Nutrition Working Group of the International Olympic Committee. (2003). "".
- Vernon, Young; Peter Pellett. (1994). "Plant proteins in relation to human protein and amino acid nutrition". American Journal of Clinical Nutrition 59 p. 1203S-l2S.
- Steinke, Waggle (1992). New protein foods in human health: nutrition, prevention and therapy CRC Press [S.l.] ISBN 978-0-8493-6904-9.
- FAO (1985). Amino acid content of foods and biological data on proteins (FAO nutritional studies number 24) Food and Agriculture Organization [S.l.] ISBN 92-5-001102-4.
- Latham, Michael C. (1997). Human nutrition in the developing world Food and Agriculture Organization of the United Nations [S.l.]
- Ten Have, GA; Engelen MP, Luiking YC, Deutz NE.. (2007). "Absorption Kinetics of Amino Acids, Peptides, and Intact Proteins". International Journal of Sport Nutrition and Exercise Metabolism 17: S23–S36. PMID 18577772.
- Food and Nutrition Board (2005). A Report of the Panel on Macronutrients, Subcommittees on Upper Reference Levels of Nutrients and Interpretation and Uses of Dietary Reference Intakes, and the Standing Committee on the Scientific Evaluation of Dietary Reference Intakes. Dietary Reference Intakes for Energy, Carbohydrate, Fiber, Fat, Fatty Acids, Cholesterol, Protein, and Amino Acids (Macronutrients) (Washington, D.C.: The National Academies Press). ISBN 0-309-08537-3.
- Elliott, Paul; Stamler, Jeremiah; Dyer, Alan R.; Appel, Lawrence; Dennis, Barbara; Kesteloot, Hugo; Ueshima, Hirotsugu; Okayama, Akira; Chan, Queenie; Garside, Daniel B.; Beifan, Zhou. (2006). "Association between protein intake and blood pressure: the INTERMAP Study". Archives of Internal Medicine 166 (1). DOI:10.1001/archinte.166.1.79. PMID 16401814.
- Otten, Jennifer J.; Jennifer Pitzi Hellwig, Linda D. Meyers (2006). Dietary Reference Intake: The Essential Guide to Nutrient Requirements (PDF) Institute of Medicine of the National Academies [S.l.]ISBN 0-309-10091-7.
- USDA (2009). Infant Nutrition and Feeding (PDF) United States Department of Agriculture [S.l.]
- biology-online.org (s. d.). «Tertiary Structure». Consultado em 8 de agosto de 2014.
- biology-online.org Dictionary (s. d. b). «Quaternary Structure». Consultado em 8 de agosto de 2014.
- Evert, Alison (2009). «Protein in diet». United States National Library of Medicine, National Institutes of Health.
- RCSB (s. d.). «RCSB Protein Data Bank». Consultado em 1 de julho de 2013.
- Organização Mundial de Saúde (2007). «Protein and amino acid requirements in human nutrition»(PDF). WHO Press. Consultado em 8 de julho de 2008.
- Institute of Medicine (2005). «Dietary reference intakes: macronutrients». Consultado em 18 de maio de 2008.
- EFSA (2012). «EFSA sets population reference intakes for protein». Consultado em 2 de setembro de 2014.
- Zeratsky, Katherine (2015.). «Are high-protein diets safe for weight loss?». Mayo Clinic. Consultado em 30 de julho de 2015.
Ligações externas[editar | editar código-fonte]
Bases de dados e projetos[editar | editar código-fonte]
- Research Collaboratory for Structural Bioinformatics - Protein Data Bank (ver também Molecule of the Month: pequenos artigos sobre proteínas selecionadas entre a base de dados)
- Protein Databank in Europe (ver também PDBeQuips: pequenos artigos e tutoriais sobre estruturas notáveis)
- NCBI Entrez Protein database
- NCBI Protein Structure database
- Human Protein Atlas
- The Protein Naming Utility
- Human Protein Reference Database
- Human Proteinpedia
- Folding@Home, Universidade de Stanford
- Comparative Toxicogenomics Database: base de dados de interações proteína-químicos, assim como relações entre genes/proteínas e doenças e relações entre químicos e doenças.
- Bioinformatic Harvester: metamotor de pesquisa (29 bases de dados) de informação sobre genes e proteínas.
- Proteopedia – Life in 3D: modelos 3D que podem ser rodados e ampliados, com anotações wiki para cada estrutura molecular proteica.
- UniProt the Universal Protein Resource
- neXtProt – Exploring the universe of human proteins: base de conhecimento sobre proteínas humanas
- MOPED Multi-Omics Profiling Expression Database: dados sobre proteínas/genes humanos e modelo
Nutrição[editar | editar código-fonte]
- Tabela nutricional com quantidade de proteína por cada 100g de alimento, Departamento de Agricultura dos Estados Unidos










Nenhum comentário:
Postar um comentário